生物适应区预测中,使用ENMTools进行冗余样点剔除
ENMTools工具筛选数据原理:
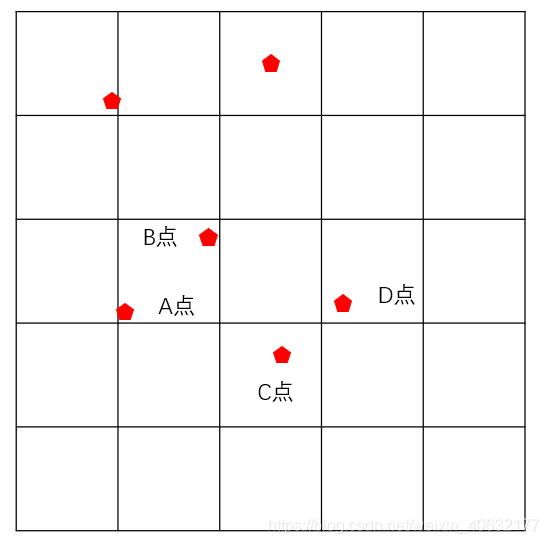
ENMTools工具可以自动匹配分析所用的环境因子栅格大小,可以删除同一栅格内的冗余数据,而不是基于距离法来删除数据。此方法快捷高效,分析结果更合理,英文期刊多用该方法。通过该方法可以删除下图中的A点或B点(同一栅格内),而C点和D点虽然直线距离小于缓冲区范围,但是不会被勿删。
引用CSDN博主「生信小窝」的说法
原文链接:https://blog.csdn.net/weixin_40632177/article/details/111502337
网上对于ENMTools这个软件或者R程序包的中文教程挺少,大多是英文文章或者原作者的视频,如果直接使用ENMTools这个软件的话,其实要方便很多,教学视频也容易找到。但使用R的教程却少之又少,我刚开始所使用的代码是B站up和谐小农民给出来的,但存在无法安装ENMTools包或运行过程报错的情况。经过GitHub原项目文档查询和debug,最终修改代码如下:
1 | install.packages("devtools") |
使用此代码,我将1164个点位减少到了324个,并且没有像使用ENMTools软件出现坐标偏移的情况。
- 标题: 生物适应区预测中,使用ENMTools进行冗余样点剔除
- 作者: Riceneeder
- 创建于 : 2023-08-30 03:53:44
- 更新于 : 2025-11-28 16:40:49
- 链接: https://gankun.cn.lu/posts/2023-08-30/
- 版权声明: 版权所有 © Riceneeder,禁止转载。
评论